Supplementary Material
A. Determination of Residue Depth cut-off values
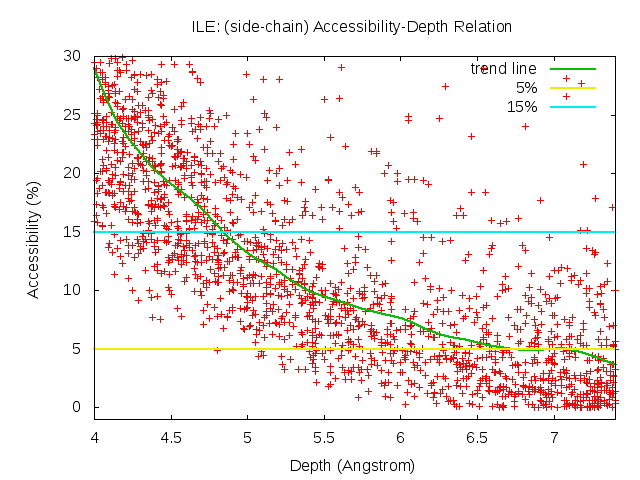
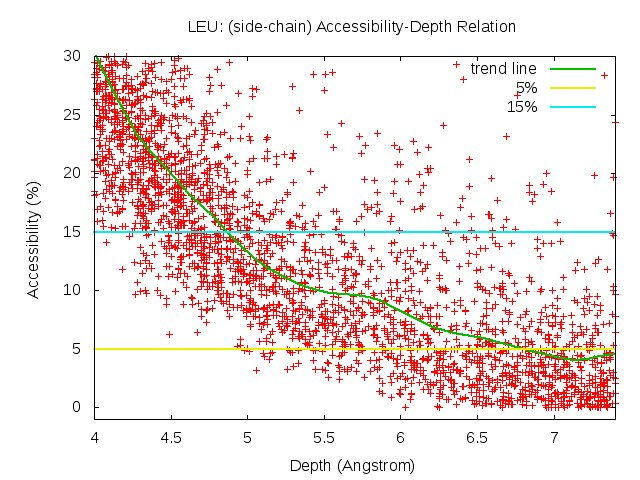
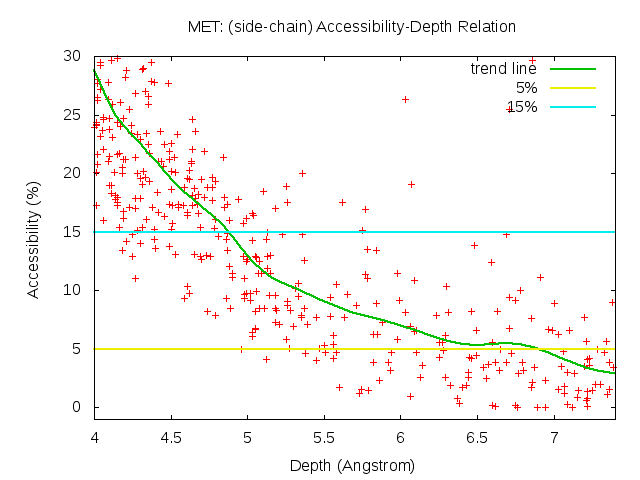
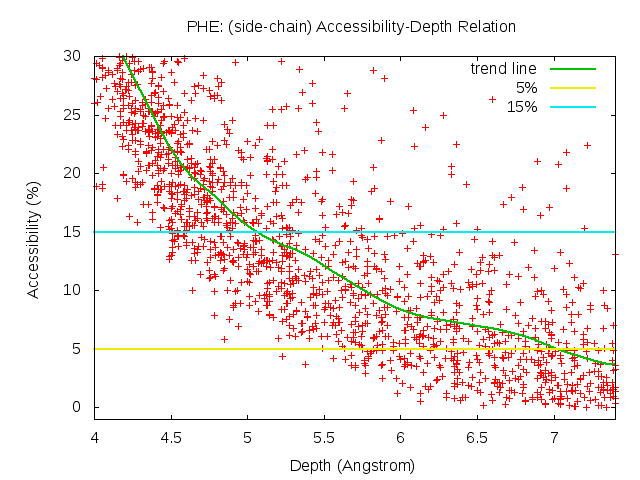
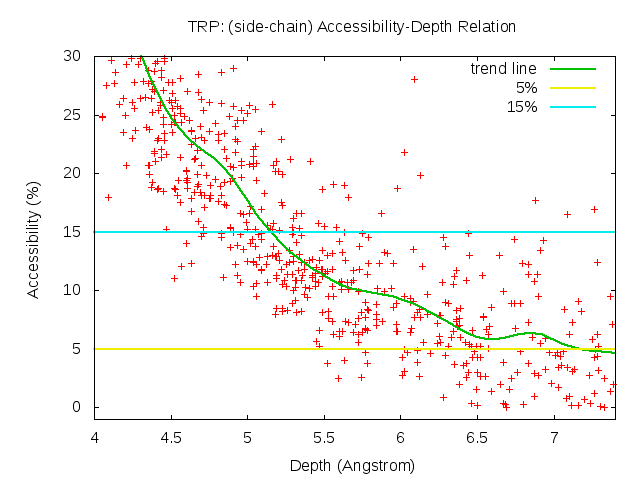
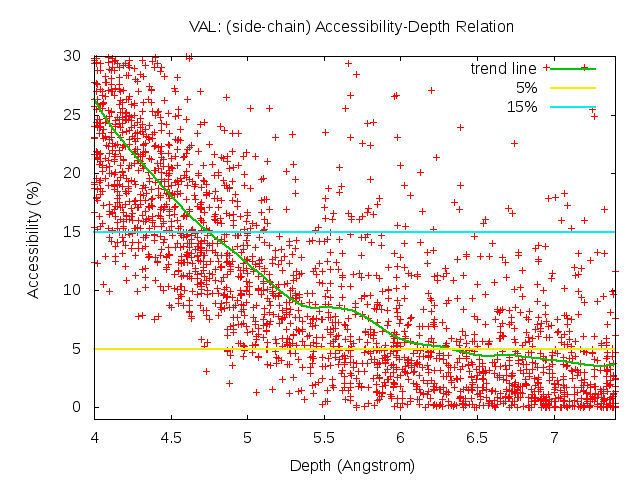
To investigate the relation between residue depth and accessibility, a training set of 561 protein structure was constructed (see main text). The values of residue depth and accessibility were computed for hydrophobic residues in this training set. A bezier regression line was fitted using the data. The depth cut-off values were then found at where the regression line intersects with accessibility of 5%.
|
Residue Type |
Number of Residues |
Cut-off values |
|
ILE |
4368 |
6.75 |
|
LEU |
6596 |
6.75 |
|
MET |
1191 |
7.00 |
|
PHE |
3251 |
7.00 |
|
TRP |
1120 |
7.00 |
|
VAL |
5681 |
6.25 |
Table A.1. Number of residues of different amino acid types and their depth cut-off values in the training set
1A6M:A, 1AQZ:A, 1B0B:A, 1B9O:A, 1BQK:A, 1C7K:A, 1CCW:A, 1CXQ:A, 1DK8:A, 1DWK:A, 1DY5:A, 1E7L:A, 1EAJ:A, 1EAQ:A, 1EB6:A, 1EUW:A, 1EXR:A, 1F4P:A, 1FJJ:A, 1FM0:E, 1G8K:B, 1GK8:I, 1GNY:A, 1GQV:A, 1GU2:A, 1GWM:A, 1H4A:X, 1H97:A, 1HFE:S, 1I76:A, 1J98:A, 1J9B:A, 1JBE:A, 1JLT:A, 1JLT:B, 1K2X:A, 1K2X:B, 1KMT:A, 1KNG:A, 1KNM:A, 1KQ6:A, 1KQR:A, 1L2H:A, 1LO7:A, 1MC2:A, 1MQK:L, 1N62:A, 1NC7:A, 1NKI:A, 1NWW:A, 1NWZ:A, 1OAL:A, 1OAQ:H, 1OAQ:L, 1OD3:A, 1OH4:A, 1OKO:A, 1OOH:A, 1OUW:A, 1P0Z:A, 1P28:A, 1P6O:A, 1PM1:X, 1PQH:A, 1PWA:A, 1QD9:A, 1QDD:A, 1QJP:A, 1QTN:A, 1QVE:A, 1R26:A, 1R29:A, 1R2Q:A, 1R8S:A, 1RG8:A, 1RM6:C, 1S69:A, 1SEN:A, 1SJW:A, 1SMB:A, 1SX7:A, 1SXV:A, 1T0A:A, 1T3Y:A, 1TO4:A, 1TT8:A, 1TU9:A, 1TYJ:A, 1U69:A, 1U7I:A, 1UFY:A, 1UGW:A, 1UNQ:A, 1UOW:A, 1UUY:A, 1UXZ:A, 1UY4:A, 1VJF:A, 1VKE:A, 1VKK:A, 1VL7:A, 1VL9:A, 1VMF:A, 1VMH:A, 1VMJ:A, 1VPM:A, 1VR7:A, 1W0N:A, 1W9S:A, 1WCU:A, 1WDD:S, 1WKQ:A, 1WLU:A, 1WMS:A, 1WMZ:A, 1WWI:A, 1X6Z:A, 1XBI:A, 1XG0:C, 1XJU:A, 1XO7:A, 1XPH:A, 1XRK:A, 1XSO:A, 1XT5:A, 1Y55:X, 1Y93:A, 1YN9:A, 1YPH:C, 1YPQ:A, 1Z2U:A, 1Z6M:A, 1ZKI:A, 1ZKK:A, 1ZLB:A, 1ZV9:A, 2A2K:A, 2A3M:A, 2A50:B, 2AEN:A, 2AJ7:A, 2ANX:A, 2AQ6:A, 2AQM:A, 2AQP:A, 2AU7:A, 2AXW:A, 2BEM:A, 2BK9:A, 2BKM:A, 2BWK:A, 2C4B:A, 2C4W:A, 2C6U:A, 2CAL:A, 2CE2:X, 2CHC:A, 2CJ4:A, 2CJT:A, 2CKK:A, 2CM5:A, 2D37:A, 2D4P:A, 2DKO:A, 2DT4:A, 2DXE:A, 2ERX:A, 2F01:A, 2F22:A, 2F62:A, 2F9S:A, 2FCL:A, 2FG1:A, 2FLH:A, 2FR5:A, 2FUF:A, 2FWH:A, 2G1U:A, 2G2S:B, 2G7B:A, 2GJ3:A, 2GKG:A, 2GLZ:A, 2GMY:A, 2GUD:A, 2GYQ:A, 2H2R:A, 2H8E:A, 2H8F:A, 2H8F:B, 2HEW:F, 2HUH:A, 2HW2:A, 2HX5:A, 2HXS:A, 2I3H:A, 2I6C:A, 2I8D:A, 2I8T:A, 2IA1:A, 2IBL:A, 2IMJ:A, 2ION:A, 2J1S:A, 2J23:A, 2J8W:A, 2JCQ:A, 2JDA:A, 2JDC:A, 2JJU:A, 2JLI:A, 2LIS:A, 2NRL:A, 2NSZ:A, 2NWF:A, 2O7A:A, 2O8Q:A, 2O90:A, 2OA2:A, 2OFC:A, 2OFZ:A, 2OH1:A, 2OIZ:D, 2OKF:A, 2OLM:A, 2OSS:A, 2OXG:Y, 2OZH:A, 2P45:B, 2P6O:A, 2P7O:A, 2P8G:A, 2PBD:P, 2PIE:A, 2PR7:A, 2PRV:A, 2Q2H:A, 2Q3T:A, 2Q4N:A, 2QCB:A, 2QEU:A, 2QF4:A, 2QF9:A, 2QIM:A, 2QIP:A, 2QJZ:A, 2QL8:A, 2QLW:A, 2QNL:A, 2QNT:A, 2QUD:A, 2R0X:A, 2R4I:A, 2R6Q:A, 2R99:A, 2RA9:A, 2RBD:A, 2RE2:A, 2RFR:A, 2RI7:A, 2RKQ:A, 2UYK:A, 2UYW:A, 2UYZ:A, 2V0U:A, 2V1M:A, 2V4X:A, 2V7F:A, 2V9V:A, 2VB1:A, 2VIF:A, 2VLQ:B, 2VMH:A, 2VUV:A, 2VVP:A, 2VXT:I, 2VY7:A, 2VZC:A, 2VZP:A, 2W1W:A, 2W3J:A, 2W72:A, 2W72:B, 2W87:A, 2WCW:A, 2WFI:A, 2WKK:A, 2WLV:A, 2WNX:A, 2WQ4:A, 2WRA:A, 2WTP:A, 2WVF:A, 2WWE:A, 2WYT:A, 2WZ8:A, 2WZ9:A, 2X2S:A, 2X32:A, 2X46:A, 2X52:A, 2X5P:A, 2X5Y:A, 2X7K:A, 2XFG:B, 2XG3:A, 2XHF:A, 2XOL:A, 2XOM:A, 2XR6:A, 2XS4:A, 2XST:A, 2Y0O:A, 2Y1Q:A, 2Y71:A, 2Y78:A, 2Y8E:A, 2Y8G:A, 2Y9F:A, 2YFU:A, 2YH5:A, 2YKZ:A, 2YZ1:A, 2Z0X:A, 2Z3H:A, 2Z51:A, 2Z6W:A, 2ZEX:A, 2ZFD:B, 2ZHN:A, 2ZIB:A, 2ZNR:A, 2ZS0:A, 2ZS0:B, 2ZS0:C, 2ZS0:D, 3A57:A, 3A6R:A, 3AP9:A, 3AQJ:A, 3AWU:B, 3B79:A, 3BA3:A, 3BED:A, 3BFQ:G, 3BLN:A, 3BS2:A, 3BT5:A, 3C3M:A, 3C8L:A, 3CE1:A, 3CHM:A, 3CI6:A, 3CIP:G, 3CJE:A, 3CM3:A, 3CP5:A, 3CT6:A, 3CTG:A, 3CXK:A, 3D0J:A, 3D1P:A, 3D22:A, 3D5P:A, 3D9X:A, 3DAU:A, 3DB7:A, 3DD7:A, 3E10:A, 3E4G:A, 3E8M:A, 3EBT:A, 3EC9:A, 3EDO:A, 3EER:A, 3EF4:A, 3EF8:A, 3EJV:A, 3EUR:A, 3EVO:A, 3EY6:A, 3EY8:A, 3F1P:B, 3F2Z:A, 3F37:A, 3F6Q:A, 3F6V:A, 3F7E:A, 3F7L:A, 3F7X:A, 3F8X:A, 3FAJ:A, 3FCN:A, 3FIA:A, 3FIQ:A, 3FK8:A, 3FQ4:A, 3FSA:A, 3FYM:A, 3FZW:A, 3G0K:A, 3G16:A, 3G39:A, 3G46:A, 3G9A:B, 3GA4:A, 3GAX:A, 3GBW:A, 3GHJ:A, 3GIX:A, 3GKM:A, 3GMX:B, 3GPG:A, 3GRD:A, 3GXH:A, 3GY9:A, 3GZB:A, 3GZR:A, 3H08:A, 3H5J:A, 3H6Q:A, 3H87:A, 3HM4:A, 3HT1:A, 3HTN:A, 3HUP:A, 3HV2:A, 3HWU:A, 3HX8:A, 3HZA:A, 3HZP:A, 3I3F:A, 3I7M:A, 3IA4:A, 3IIS:M, 3IJM:A, 3IMK:A, 3IP0:A, 3IQ1:A, 3IQ2:A, 3IQT:A, 3ITF:A, 3IVV:A, 3JRV:A, 3JUD:A, 3JVL:A, 3K67:A, 3KA8:A, 3KBE:A, 3KE7:A, 3KFF:A, 3KHQ:A, 3KKG:A, 3KLR:A, 3KUU:A, 3KWK:A, 3KWU:A, 3KYZ:A, 3L0F:A, 3L0F:B, 3L4R:A, 3L51:A, 3L51:B, 3L8H:A, 3L91:A, 3LAS:A, 3LB2:A, 3LBE:A, 3LHN:A, 3LJD:A, 3LJW:A, 3LLO:A, 3LQW:A, 3LWZ:A, 3LX3:A, 3LYD:A, 3LYG:A, 3LZL:A, 3M1H:A, 3M1O:A, 3M1X:A, 3M2O:A, 3M7A:A, 3MC3:A, 3MDX:A, 3ML1:B, 3MMH:A, 3MNG:A, 3MPZ:A, 3MR0:A, 3N08:A, 3N1F:A, 3N4I:B, 3N4J:A, 3N5A:A, 3NBC:A, 3NBK:A, 3NEC:A, 3NJK:A, 3NKV:A, 3NOH:A, 3O1C:A, 3O2R:A, 3O5Q:A, 3OBL:A, 3OD9:A, 3OGN:A, 3OHE:A, 3OJ0:A, 3OJ6:A, 3OMD:A, 3ON9:A, 3ONH:A, 3OR5:A, 3OSE:A, 3OST:A, 3OXP:A, 3P1G:A, 3P3V:A, 3P48:A, 3P6D:A, 3P6I:A, 3PG6:A, 3PIW:A, 3PJY:A, 3PNA:A, 3PP2:A, 3PWF:A, 3Q62:A, 3Q64:A, 3QDS:A, 3QHB:A, 3QL9:A, 3QOO:A, 3QU3:A, 3QZB:A, 3QZM:A, 3R0L:D, 3R0N:A, 3R2R:A, 3R3Q:A, 3RDO:A, 3ROB:A, 3ROF:A, 3RRI:A, 3RWN:A, 3S4K:A, 3S6F:A, 3S6M:A, 3S8I:A, 3S9X:A, 3SB1:A, 3SK2:A, 3SM1:A, 3SXH:A, 3T3L:A, 3TQ5:A, 3ZW5:A, 3ZXH:A, 4A2V:A, 4UBP:B
Table A.2. PDB ID of proteins in the training set






Figure A.1. Residue depth – Accessibility relation of different amino acid types in the training set. Bezier regression line was colored in green and the 5% accessibility was represented as a horizontal line colored in yellow.
B. Temperature sensitive position predictions without experiment validations
|
|
|
|
|
|
|
|
|
|
Protein |
PDB ID |
Chain |
Residue |
Residue |
Seq. based |
Structure based |
Predicted by |
|
|
|
length |
position |
Type |
method |
method |
both |
|
gene V |
1YHA |
87 |
4 |
VAL |
− |
√ |
− |
|
|
|
|
44 |
LEU |
√ |
− |
− |
|
lambda repressor |
1LMB |
92 |
69 |
LEU |
− |
√ |
− |
|
|
|
|
73 |
VAL |
− |
√ |
− |
|
|
|
|
68 |
ILE |
√ |
− |
− |
|
T4 lysozyme |
2LZM |
164 |
7 |
LEU |
√ |
√ |
√ |
|
|
|
|
27 |
ILE |
− |
√ |
− |
|
|
|
|
29 |
ILE |
− |
√ |
− |
|
|
|
|
46 |
LEU |
− |
√ |
− |
|
|
|
|
78 |
ILE |
− |
√ |
− |
|
|
|
|
84 |
LEU |
− |
√ |
− |
|
|
|
|
99 |
LEU |
− |
√ |
− |
|
|
|
|
111 |
VAL |
− |
√ |
− |
|
|
|
|
121 |
LEU |
− |
√ |
− |
|
|
|
|
133 |
LEU |
− |
√ |
− |
|
|
|
|
4 |
PHE |
√ |
− |
− |
|
|
|
|
87 |
VAL |
√ |
− |
− |
|
|
|
|
100 |
ILE |
√ |
− |
− |
|
|
|
|
104 |
PHE |
√ |
− |
− |
|
Gal4 |
3COQ |
88 |
64 |
LEU |
√ |
√ |
√ |
|
|
|
|
67 |
LEU |
− |
√ |
− |
|
|
|
|
71 |
ILE |
√ |
− |
− |
|
|
|
|
72 |
PHE |
√ |
− |
− |
|
|
|
|
80 |
ILE |
√ |
− |
− |
|
Ura3 |
1DQW |
267 |
21 |
LEU |
√ |
√ |
√ |
|
|
|
|
22 |
PHE |
√ |
√ |
√ |
|
|
|
|
48 |
VAL |
√ |
√ |
√ |
|
|
|
|
51 |
LEU |
√ |
√ |
√ |
|
|
|
|
58 |
LEU |
√ |
√ |
√ |
|
|
|
|
65 |
LEU |
√ |
√ |
√ |
|
|
|
|
88 |
LEU |
√ |
√ |
√ |
|
|
|
|
89 |
PHE |
√ |
√ |
√ |
|
|
|
|
125 |
VAL |
√ |
√ |
√ |
|
|
|
|
148 |
LEU |
√ |
√ |
√ |
|
|
|
|
149 |
MET |
√ |
√ |
√ |
|
|
|
|
179 |
VAL |
√ |
√ |
√ |
|
|
|
|
180 |
ILE |
√ |
√ |
√ |
|
|
|
|
182 |
PHE |
√ |
√ |
√ |
|
|
|
|
198 |
LEU |
√ |
√ |
√ |
|
|
|
|
199 |
ILE |
√ |
√ |
√ |
|
|
|
|
200 |
MET |
√ |
√ |
√ |
|
|
|
|
231 |
ILE |
√ |
√ |
√ |
|
|
|
|
233 |
VAL |
√ |
√ |
√ |
|
|
|
|
17 |
VAL |
− |
√ |
− |
|
|
|
|
36 |
LEU |
− |
√ |
− |
|
|
|
|
44 |
LEU |
− |
√ |
− |
|
|
|
|
55 |
ILE |
− |
√ |
− |
|
|
|
|
57 |
LEU |
− |
√ |
− |
|
|
|
|
62 |
VAL |
− |
√ |
− |
|
|
|
|
64 |
ILE |
− |
√ |
− |
|
|
|
|
77 |
LEU |
− |
√ |
− |
|
|
|
|
86 |
PHE |
− |
√ |
− |
|
|
|
|
94 |
PHE |
− |
√ |
− |
|
|
|
|
101 |
VAL |
− |
√ |
− |
|
|
|
|
112 |
ILE |
− |
√ |
− |
|
|
|
|
115 |
TRP |
− |
√ |
− |
|
|
|
|
124 |
VAL |
− |
√ |
− |
|
|
|
|
129 |
ILE |
− |
√ |
− |
|
|
|
|
133 |
LEU |
− |
√ |
− |
|
|
|
|
147 |
LEU |
− |
√ |
− |
|
|
|
|
197 |
TRP |
− |
√ |
− |
|
|
|
|
204 |
VAL |
− |
√ |
− |
|
|
|
|
223 |
VAL |
− |
√ |
− |
|
|
|
|
224 |
VAL |
− |
√ |
− |
|
|
|
|
230 |
ILE |
− |
√ |
− |
|
|
|
|
237 |
LEU |
− |
√ |
− |
|
|
|
|
87 |
LEU |
√ |
− |
− |
|
|
|
|
130 |
VAL |
√ |
− |
− |
|
|
|
|
150 |
LEU |
√ |
− |
− |
|
|
|
|
183 |
ILE |
√ |
− |
− |
|
|
|
|
232 |
ILE |
√ |
− |
− |